Nieuwe methode die de diagnose van kanker radicaal verandert Max Planck Society
Deep Visual Proteomics biedt celspecifieke, op eiwitten gebaseerde informatie voor kankeranalyse
Hoe ontstaat kanker? Hoe verandert de cellulaire structuur van een tumor zijn kwaadaardige eigenschappen? Deze vragen zijn noodzakelijk en moeilijk te beantwoorden. Ze zijn echter essentieel om kanker te begrijpen en een permanente behandeling te vinden. Een Duits-Deens team onder leiding van Matthias Mann heeft een baanbrekende technologie ontwikkeld, “Deep Visual Proteomics”. Deze technologie voorziet onderzoekers en clinici van op eiwitten gebaseerde informatie en helpt kankers nauwkeurig te begrijpen per individuele celtype. De technologie laat zijn potentie zien in een voor het eerst toepassing op kankercellen.
© MPI Biochemie
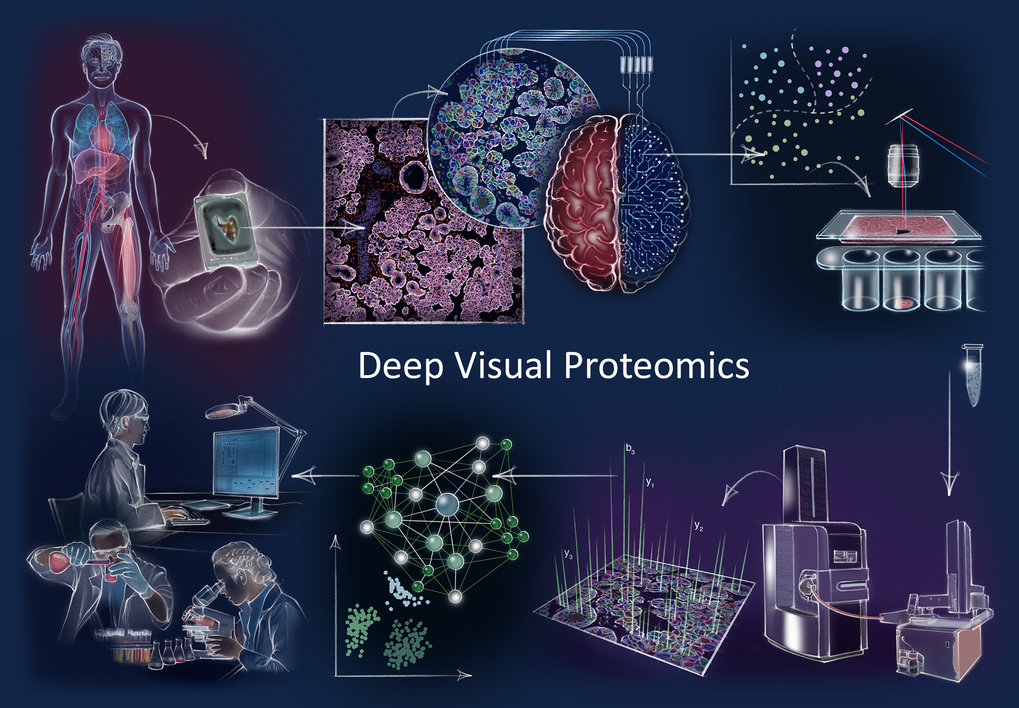
Deep Visual Proteomics-concept en workflow (met de klok mee): Deep Visual Proteomics combineert beeldvorming met hoge resolutie en AI-ondersteunde beeldanalyse om afzonderlijke cellen te classificeren en te isoleren met een nieuwe workflow voor zeer gevoelige eiwitten. Deze methode combineert data-intensieve beeldvorming van celculturen van patiënten of gearchiveerde biobankweefsels met op deep learning gebaseerde celsegmentatie en op machine learning gebaseerde identificatie van celtypen en toestanden. (Niet) gecontroleerde AI-gelabelde cellulaire of subcellulaire organismen van belang worden onderworpen aan geautomatiseerde lasermicrodissectie en op massaspectrometrie gebaseerde proteomics-profilering. Daaropvolgende bio-informatische gegevensanalyse maakt het mogelijk eiwitsignaturen vast te leggen die moleculaire inzichten verschaffen in proteomische veranderingen in gezondheids- en ziektetoestanden op eencellig niveau. © MPI Biochemie
Eiwitten zijn essentiële puzzelstukjes voor een verscheidenheid aan ziekten. Ze worden ook wel de “moleculaire werkpaarden van de cel” genoemd. De juiste functie ervan bepaalt de functie van de cel en dus ook de functie van het individu. Matthias Mann legt uit: “Als iets in onze cellen niet goed werkt en we ziek zijn, kun je er zeker van zijn dat de eiwitten op verschillende manieren betrokken zijn. Daarom kan het in kaart brengen van de aard van het eiwit ons helpen ontdekken : Waarom kon de tumor zich bij een bepaalde patiënt ontwikkelen?” Wat zijn de zwakke punten van deze tumor en welke behandelmethode is gunstig?” legt Matthias Mann uit Max Planck Instituut voor Biochemie Dicht bij München en van Novo Nordisk Proteïne Onderzoekscentrum aan de Universiteit van Kopenhagen in Denemarken.
Geïnspireerd door deze vragen ontwikkelde een multidisciplinair onderzoeksteam onder leiding van Matthias Mann een nieuwe en innovatieve methode. In de studie zullen de visuele kenmerken van de tumor worden bepaald met behulp van diepe profileringstechnologie om eiwitten te analyseren in groepen abnormale cellen die grenzen aan omliggende gezonde cellen. Deze benadering zou onderzoekers een ongekend inzicht in kanker kunnen geven en oncologen kunnen helpen bij het creëren van gerichte strategieën voor diagnose en behandeling.
Deep Optical Proteomics combineert vier technologieën
Deep Optical Proteomics combineert voor het eerst de voordelen van vier verschillende technologieën in één methodologie. Ten eerste maakt moderne microscopie weefselkaarten met een hoge resolutie. Ten tweede worden algoritmen voor machinaal leren en kunstmatige intelligentie gebruikt om cellen te classificeren op basis van hun vorm, grootte of eiwitlokalisatie voordat individuele cellen worden verzameld met behulp van lasermicrodissectie met hoge resolutie. Ten derde, nadat normale of diverse zieke celpopulaties zijn gesorteerd, worden duizenden eiwitten binnen deze celpopulaties gelijktijdig geïdentificeerd met behulp van ultragevoelige massaspectrometrie. Ten vierde genereren complexe bio-informatica-analyses eiwitkaarten die ruimtelijke resolutie van eiwitten mogelijk maken bij zeer complexe ziekten zoals kanker. Deze eiwitkaarten zijn waardevolle hulpmiddelen voor clinici om de mechanismen van gezondheid en ziekte beter te begrijpen.
“Ons nieuwe concept van ‘diepe optische proteomics’ zou een paradigmaverschuiving kunnen worden voor moleculaire pathologie in de kliniek. Met deze methode nemen we een weefselmonster met kankercellen en kunnen we in zeer korte tijd en met minimale inspanning duizenden eiwitten identificeren. Deze proteomische analyses onthullen de mechanismen die de evolutie aansturen. Nieuwe therapeutische doelen kunnen dus direct worden afgeleid uit een enkele weefselsectie van de biopsie van een patiënt”, zegt Andreas Mund, universitair hoofddocent bij het Center for Protein Research en onderdeel van het team onder leiding van Matthias Mann, die deze ontwikkeling leidde bij het Center for Protein Research en het Max Planck Institute for Biochemistry.
Relevantie voor klinische pathologie
In de studie konden de onderzoekers “diepe optische eiwitten” toepassen op cellen van patiënten met speekselklier- en huidkankerpatiënten. Lise Mette Rabek Gerderm, Consultant Consultant en Associate Professor of Clinical Research in de Department of Pathology van Sealand University Hospital Roskilde en de Department of Clinical Medicine van de University of Copenhagen beschrijft: “Deze unieke methode combineert weefselstructuuranalyse en proteomics-analyse, wat essentieel voor geselecteerde geselecteerde cellen Diagnose van een klinisch zeer complexe aandoening met behulp van Deep Visual Proteomics-analyse.
Fabian Coscia, een van de eerste auteurs van de studie gepubliceerd in Nature Biotechnology en hoofd van de onderzoeksgroep “Spatial Proteomics” sinds juni 2021 Max Delbrück Centrum voor Moleculaire Geneeskunde bij de Helmholtz Society In Berlijn zegt hij: “De technologie kan op een vergelijkbare manier ook worden gebruikt om andere soorten tumoren te beschrijven.” Het doel is om met gearchiveerde gegevens van biobanken nieuwe aanvalspunten te ontdekken voor individuele kankertherapieën en dus behandelvormen die zijn toegesneden op patiënten – en ook op voorheen therapieresistente tumoren.
En het is niet alleen kanker die beter kan worden begrepen met behulp van diepe visuele eiwitten. De methodiek kan ook worden toegepast op andere ziekten. “Je kunt bijvoorbeeld de eiwitten van een neuron analyseren om te zien wat er precies in de cel gebeurt tijdens het beloop van neurodegeneratieve ziekten zoals de ziekte van Alzheimer of de ziekte van Parkinson”, vervolgt Coscia. “Door microscopie, kunstmatige intelligentie en zeer gevoelige op massaspectrometrie gebaseerde eiwitten te combineren, hebben we een zeer krachtige methode ontwikkeld om de moleculaire circuits van gezonde en zieke cellen te begrijpen, wat clinici kan helpen bij het identificeren van medicijndoelen en toekomstige diagnostiek”, legt Matthias Mann uit. .

“Analist. Schepper. Zombiefanaat. Fervente reisjunkie. Popcultuurexpert. Alcoholfan.”


